Texto: Bruno Vaiano
Design: Natalia Sayuri
Ilustrações: Fernando Molina
Na virada do milênio, enquanto o Projeto Genoma Humano dava os toques finais no sequenciamento do nosso DNA, o biólogo Ewan Birney organizou um bolão internacional digno de Copa do Mundo. Custava um dólar por inscrição, e a pergunta era: quantos genes vamos encontrar? Nerds do mundo todo apostaram algo entre 30 mil e 120 mil.
Eram chutes. Bem informados – mas ainda assim chutes. Por volta do ano 2000, “a maioria dos compêndios estabelecia o número de 100 mil genes por nenhuma outra razão senão a de ser um conveniente número inteiro”, escreve Kevin Davies no livro Decifrando o Genoma.
Duas décadas e muitos artigos científicos depois, hoje sabemos que a cifra é mais humilde: seres humanos têm só 21 mil genes. Para fins de comparação, uma bactéria E. coli, comum em bancadas de laboratório e no seu intestino, tem 4,4 mil – aproximadamente um quarto do total humano. O genoma dela foi publicado em 1997, pouco antes do nosso, em 2001.
A bactéria tem menos. Mas não muito menos. Até então, havia uma expectativa – em parte lógica, em parte narcisista – de que seres multicelulares complexos teriam uma quantidade esmagadoramente grande de genes em comparação aos procariontes, criaturinhas microscópicas de uma célula só. Mas não é bem assim: o jeito como os genes interagem importa muito mais que o número de genes em si.
O maior mistério calhou de ser outro: a quantidade total de moléculas de DNA que carregamos é muito maior. Mais precisamente, 650 vezes maior. São 3 bilhões de letrinhas de código genético num Homo sapiens contra apenas 4,6 milhões em uma E. coli.
Traduzindo: é como se nosso caderno de anotações contivesse apenas quatro vezes mais informação que o das bactérias (os genes em si), mas essa informação ocupasse 650 vezes mais páginas (as moléculas de DNA, que são o formato de arquivo em que ficam salvos os genes). Para que tanto espaço?

Bem-vindo ao DNA lixo
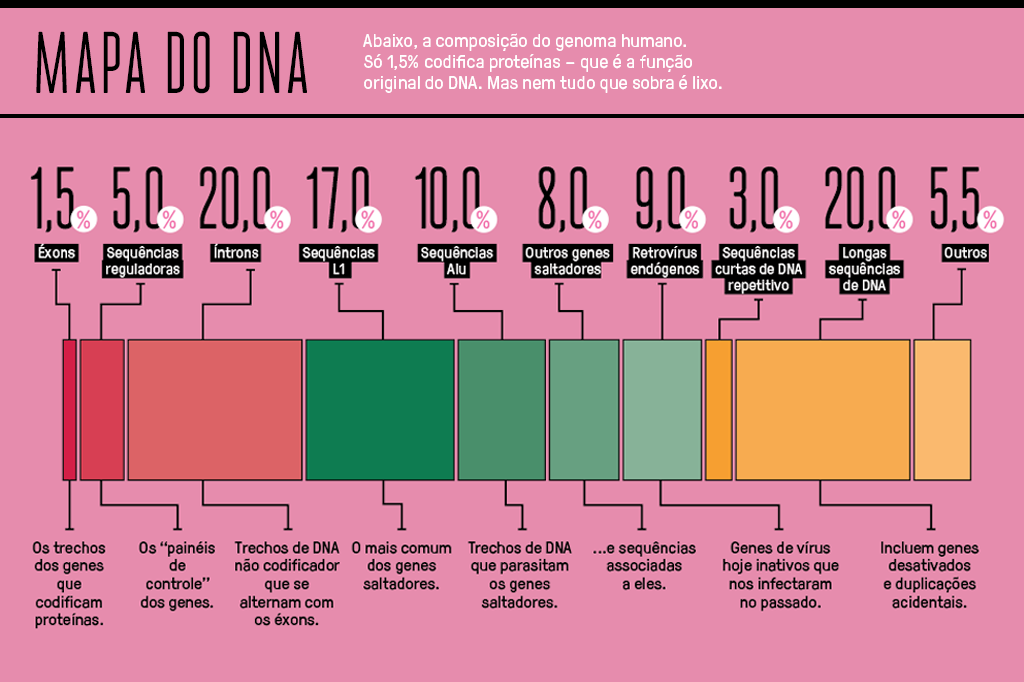
O lance é que os genes de verdade ocupam apenas 1,5% do total de DNA que um ser humano carrega. Só usamos para valer uma parcela ínfima do armazenamento total disponível em nosso cartão de memória biológico. Outros 5% consistem nas chamadas regiões reguladoras, que servem como painéis de controle para aumentar, reduzir, iniciar ou interromper a atividade de cada gene.
Os 93,5% de DNA que sobram nessa conta permanecem em sua maioria enroladinhos no núcleo das células, numa forma adormecida chamada heterocromatina. Essa porcentagem, batizada na letra fria do jargão como “DNA não codificador”, ganhou o apelido carinhoso de DNA lixo – ainda que nem tudo ali seja, de fato, inútil, o que faz alguns especialistas torcerem o nariz para o termo.
Esse novelo é um matagal molecular aleatório e relativamente inexplorado. Uma mistura de caixa de spam com ferro velho habitada por carcaças de genes aposentados, vírus antiquíssimos que se incorporaram a nós e trechos de código desordeiros que saltam de cromossomo em cromossomo, criando cópias de si mesmos em escala industrial. Existe até um consórcio de universidades americanas, chamado Encode, que se dedica a mapear – e compreender melhor – tudo isso.
Carcaças de genes e vírus aposentados: o genoma é um armário bagunçado.
Essa mistura de deep web genômica com sítio arqueológico microscópico nos dá um vislumbre de 4 bilhões de anos de evolução, e também é um palco onde a seleção natural atua no seu nível fundamental: genes que se multiplicam às nossas custas prevalecem sobre aqueles que topam a difícil empreitada de construir nossos corpos.
A deep web genética
A parte mais civilizada do DNA não codificador são os chamados íntrons, e por eles começamos a nossa jornada. Você talvez lembre que a molécula de DNA é feita enfileirando pecinhas químicas chamadas adenina (A), timina (T), guanina (G) e citosina (C) como miçangas em um colar. A ordem em que essas miçangas aparecem é um código que, ao ser lido pelo maquinário da célula, explica a ela como fabricar certas proteínas: anticorpos, enzimas digestivas, alguns hormônios, a queratina dos seus cabelos etc.
Um corpo humano exige 92 mil tipos de proteína para funcionar, e cada gene é a receita de uma delas, mas nós só temos 21 mil genes. Por que a conta não fecha? O segredo é que 90% dos nossos genes na verdade usam uma forma peculiar de compactação de arquivo para guardar a receita de duas ou mais proteínas ao mesmo tempo.
Pense numa proteína como um carro. Ela tem vários componentes: chassi, motor, transmissão, radiador. Dois carros de modelos diferentes, mas feitos pelo mesmo fabricante, geralmente compartilham alguns componentes – algo que facilita a logística e barateia a produção.
De forma similar, cada gene humano se divide em aproximadamente dez éxons: trechinhos de DNA que codificam componentes de proteína. Combinando esses componentes, dá para montar mais de uma proteína no mesmo gene.
O pulo do gato é que os éxons são separados por íntrons: enormes trechos de DNA cujas letrinhas não significam nada. Essas sequências aleatórias são só uma espécie de preenchimento que separa os éxons. Divisórias entre os setores da fábrica.
20% do conteúdo total do seu genoma são íntrons que separam os 1,5% de éxons. Adicione a isso 5% de regiões reguladoras e temos 25% do DNA humano sem codificar nada, servindo de burocracia para os 1,5% que põe a mão na massa. O resultado disso é que um gene humano tem em média 27 mil letrinhas de comprimento, contra mil das bactérias – que seguem à risca a regra de uma proteína por gene.
Achou parecido com uma repartição pública? Calma: a gente ainda nem abriu o almoxarifado.

A volta dos que não foram
A maioria dos genes atua por caminhos bioquímicos tortuosos. Cada proteína interfere na ação de inúmeras outras proteínas, gerando reações em cadeia dificílimas de decifrar. Às vezes, porém, um pedacinho de DNA mostra abertamente a que veio. É o caso do NOTCH2NL. Em 2017, um grupo de pesquisadores da Universidade da Califórnia descobriu que carregamos três cópias do dito-cujo. Tire uma, o bebê nasce com microcefalia. Adicione uma e acontece o oposto: o cérebro fica grande demais para caber dentro do crânio.
O curioso é que esse gene também existe nos chimpanzés, mas em uma versão inativa, com um erro no código. Em algum ponto da evolução humana, esse erro foi corrigido ao acaso, por uma mutação aleatória. O gene voltou a expressar sua proteína e deu um gás no sistema nervoso, permitindo a evolução de um traço típico da nossa espécie: o cabeção.
É uma história comum. 15% do nosso DNA consiste em fragmentos de genes e pseudogenes. São trechos de DNA claramente reconhecíveis como receitas de proteína, mas incapazes de se expressar. Esses genes podem ser cópias acidentais que vieram sem as regiões reguladoras – ou seja: sem os botões que permitiriam acioná-los. Outros acumularam muitas mutações e acabaram caindo no ostracismo, incapazes de realizar suas funções originais. Um único gene fantasma no cromossomo 9 tem 14 cópias enferrujadas, espalhadas como Titanics naufragados pelo núcleo das células.
Por que essa bagunça se acumula? “Há alguns anos eu percebi que há dois tipos de porcarias no mundo, e que a maioria das línguas têm palavras diferentes para distingui-las”, escreveu o geneticista Sidney Brenner em 1998. “Existem as porcarias que você guarda, que são tralhas, e as porcarias que você joga fora, que são lixo. O DNA em excesso nos nossos genomas é tralha, e está lá porque é inofensivo – e porque os processos moleculares gerando DNA extra são mais rápidos que os processos que se livram dele.”
Elementos transpositores
A tralha pode até ser inofensiva, mas isso não significa que não possa ser incômoda. Todo ser humano carrega em seu genoma umas 100 mil exemplares de trechinhos de DNA chamados LINE-1 (ou, simplesmente, L1). Um L1 comum tem 6 mil letrinhas de comprimento. É tanto L1 – mas tanto L1 – que 17% de todo o nosso material genético consiste nesses carinhas.
O L1 contém o gene para produzir uma proteína chamada transcriptase reversa. Essa proteína participa de um processo que gera uma cópia do L1 e então instala essa cópia de volta no genoma. Uma vez instalada, a cópia pode gerar uma cópia da cópia. Que então vai gerar uma cópia da cópia da cópia. O L1 é como uma impressora que imprime impressoras. Seu fim é si mesmo. O que explica como tantos deles foram parar dentro de nós.
Elementos transpositores que fazem “copiar e colar” são chamados de retrotransposons. Eles são mais comuns. Mas também existem os transposons, que fazem “recortar e colar” – não deixam uma cópia no local de origem. Eles só viajam de cromossomo em cromossomo. Em qualquer um dos casos, esses genes saltadores (como foram apelidados) corrompem a cadeia de DNA no local em que se inserem.
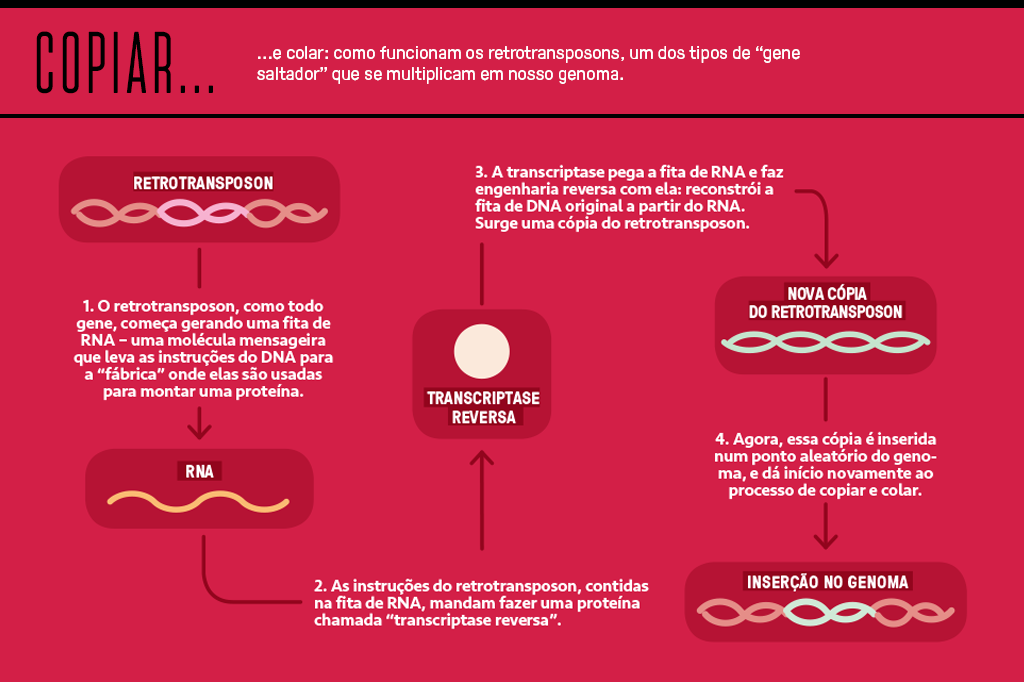
Nós aturamos 100 mil cópias de um pedacinho de DNA que bugou – e cria cópias de si mesmo com o ímpeto de uma máquina de xerox.
Todo malandro tem seu mané. O L1 usa a proteína que você come para fazer mais de si mesmo – o que já é sacana –, mas o interessante é que ele próprio tem seus parasitas, chamados Alus. Esses são trechinhos de DNA ainda menores e ainda mais numerosos, com algo entre 180 e 280 letrinhas de comprimento. Em vez de fabricarem a própria transcriptase reversa (eles são curtos demais para guardar a receita), os Alus roubam a transcriptase dos L1s. Por esse método, conseguiram acumular mais de um milhão de cópias no nosso genoma – e no de nossos primos chimpanzés, já que esse pequeno elemento entrou na nossa linhagem antes de nos separarmos deles.
Existem sequências ainda mais curtinhas e repetitivas que os Alus, chamadas minissatélites hipervariáveis. Elas se embaralham como cartas na mão de um crupiê, e cada um de nós tem uma impressão digital única formada por essas letrinhas. São elas que os cientistas forenses usam para desvendar crimes.
Barbara McClintock, filha rebelde de uma família ultraconservadora que fez faculdade contra os desejos da mãe, descobriu os genes saltadores em 1936 pesquisando espigas de milho. No Brasil, só conhecemos bem as espigas amarelas, mas você já deve ter visto – talvez em um programa de TV sobre o México – espigas com grãos multicoloridos: brancos, roxos, pretos, alguns pontilhados como dálmatas.
McClintock percebeu que essas variações de pigmentação acontecem quando genes saltadores caem no meio dos genes para pigmentação, causando disrupções aleatórias por toda a espiga. O milho sofre de uma infestação que faz a nossa parecer um passeio no parque: 85% do genoma da espécie consiste em retrotransposons. De fato, em comparação com outras espécies, os humanos são razoavelmente livres desses bugs no código genético. Só uma em cada 700 mutações nos nossos genomas é culpa de genes saltadores. Em camundongos de laboratório, é uma em dez.
Sabe-se que os elementos transpositores, quando se livram de suas algemas, bagunçam o genoma e colaboram com tumores e outras doenças. Porém, mutações não são só sinônimo de problema. Também são sinônimo de variedade. E variedade é justamente o combustível da seleção natural. Uma hipótese popular na neurociência é que a dança das cadeiras propiciada pelos genes saltadores, nos nossos neurônios, seja responsável pela variabilidade entre cérebros típica da nossa espécie – o que já é, só neste texto, o segundo exemplo de DNA lixo sendo crucial para a evolução humana.
Às vezes, um gene saltador pula de uma espécie para outra de carona em um vetor. As moscas Drosophila melanogaster, organismos-modelo criados em laboratórios do mundo todo para pesquisas em genética, já sofreram uma infestação desse gênero, oriunda de uma mosca selvagem. Hoje, a situação é estável, mas o genoma desses insetos ficou lotado dos chamados “elementos P”.
De maneira similar, um gene saltador inofensivo em outro animal poderia se espalhar entre seres humanos com consequências imprevisíveis caso realizássemos xenotransplantes: a troca de órgãos ou tecidos entre duas espécies. A ideia de zerar a fila por rins ou corações retirando-os de porcos ou primatas apareceu na literatura científica em 1905 e permanece uma possibilidade teórica (ou quase isso: houve experimentos formidavelmente antiéticos no começo do século 20, e em 1984 uma criança americana sobreviveu 21 dias com um coração de babuíno até rejeitar o órgão. Não havia um coração humano disponível).
Com o barateamento da técnica Crispr de edição de DNA, a ideia para as próximas décadas é editar os animais doadores para criá-los sem genes saltadores nem incompatibilidades com o sistema imunológico.
–

Genes em carreira solo
Aqui há um problema de ovo e galinha. Ninguém sabe se os vírus evoluíram a partir de elementos transpositores – que teriam desenvolvido a capacidade de saltar de ser vivo em ser vivo – ou se aconteceu o contrário: os elementos transpositores é que seriam descendentes de vírus, que se simplificaram e estabeleceram moradia definitiva no genoma.
Os retrovírus – cujo exemplo mais famoso é o HIV – usam a transcriptase reversa para se instalar no genoma das células do hospedeiro, exatamente como faz o L1. Sinal de que esses seres peculiares podem compartilhar algum grau de parentesco. Quando um retrovírus infecta uma célula da linha germinativa (ou seja, uma célula que vai virar um espermatozoide ou óvulo) o material genético do parasita acaba indo de brinde em todas as células do bebê. Graças a esse processo, 8% do genoma humano hoje consiste em retrovírus endógenos, conhecidos pela sigla HERVs.
Nem todos estão inativos. Há um conhecido conjunto de genes de origem viral que se expressa abundantemente na placenta, codificando proteínas importantes para a gestação. O DNA pode até ser lixo, mas ele é reciclável – e nossa espécie tirou muito código genético do ostracismo ao longo da evolução.
A capacidade de reaproveitar genes fornecidos por outras espécies é rara em nós, mas comum entre bactérias. Essas criaturinhas unicelulares possuem apenas um cromossomo, que tem forma de anel. Em torno dele, flutuando languidamente no citoplasma, distribuem-se pedacinhos de DNA nos chamados plasmídeos, que contêm genes hippies caroneiros: pulam de bactéria em bactéria fornecendo receitas de proteínas novas para quem quiser adotá-las.
O nome desse troca-troca de plasmídeos é transferência horizontal de genes, e ele talvez seja o maior perigo de saúde pública do século 21, pois permite que as bactérias criem resistência a antibióticos rapidamente. Ao incorporar esses genes autônomos à equipe, os micróbios obtêm o passo a passo para montar antídotos contra as drogas que usamos para matá-los.
Para as bactérias, a evolução não é um processo vertical – em que uma geração vem após a outra, e todos morrem como nasceram. O fenômeno da vida se torna um intercâmbio eterno de repertório genético, e identidade é um conceito fluido: uma bactéria pode se tornar outra absorvendo e incorporando seus genes. A flexibilidade é tanta, de fato, que vale enquadrar a cena de outra perspectiva: a Terra primitiva era um mundo de genes, alguns optando por viver em bando, outros fazendo carreira solo.
A ideia de que há conflito e cooperação dentro do nosso genoma se torna natural quando paramos de pensar nos genes como um cartão de memória a serviço do nosso corpo e invertemos o ponto de vista: na verdade, o corpo é uma criação dos genes.
Eles são moléculas inanimadas, é claro, e essas relações são meramente químicas. Não há intenções, boas ou más. Suponha, por exemplo, que uma mutação aleatória no cromossomo Y dê origem a um gene que boicota espermatozoides com o cromossomo X. (Lembre-se da regra básica: se o espermatozoide que fecunda o óvulo é Y, nasce um menino; se ele é X, nasce uma menina. O óvulo é sempre X, é o homem que “decide” o sexo dos bebês.)
Em poucas gerações, esse gene vai destruir o equilíbrio numérico entre os sexos. Vão nascer mais meninos do que meninas, e esses meninos virão equipados com o gene criador de mais meninos. O processo só vai parar se surgir do nada algum mecanismo capaz de amordaçar esse gene.

Essa não é só uma possibilidade teórica. É algo que acontece – e explica por que o cromossomo Y é bem menor que o X. Ao longo de milhões de anos de seleção natural, a maior parte dos genes do cromossomo Y foi amarrada numa camisa de força de heterocromatina, à prova de rebeliões. Apenas alguns poucos genes, os realmente essenciais para a masculinização dos fetos, são autorizados a se expressar. Hoje, o cromossomo X tem 1.600 genes funcionais. O Y, 50. A maior parte do Y virou DNA não codificante. DNA lixo, aposentado na marra.
Apesar de tanto conflito, todas as nossas células topam se sacrificar e morrer conosco – para que apenas uma célula, o espermatozoide ou o óvulo, dê origem a um bebê. É um fato mágico que, a cada geração, nosso genoma pega precisamente metade de toda a sua colossal tralha, põe num caminhão de mudança e constrói um ser humano novo do zero, com uma ajudinha da tralha de outra pessoa. Cada filhote traz uma nova sociedade de genes, talvez mais bem equipada para o mundo lá fora, talvez menos.
A cada geração, nosso genoma empacota metade de sua tralha em uma gameta e faz um novo bebê – com uma ajudinha da tralha de outra pessoa.
Por que nossos genes não declaram independência e ganham a vida se multiplicando sozinhos, sem ter de reproduzir todo o maquinário de um ser vivo completo? Bem, eles declaram: os L1s, Alus, plasmídeos e até os vírus estão aí para provar. Por que nossas células não fazem o mesmo? Bem, às vezes elas fazem: os tumores são pedaços do nosso corpo que crescem às nossas custas em vez de colaborar conosco. Mesmo assim, estamos aqui – e todos os outros animais, plantas e demais espécies também. Sinal de que a união, na natureza, às vezes é a saída mais vantajosa para todos os envolvidos. Se não fosse, a seleção natural já teria dado um jeito nisso.

Fontes: livros Decifrando o Genoma, de Kevin Davies; Genome, de Matt Ridley; The Red Queen de Matt Ridley; The Extended Phenotype, de Richard Dawkins; The Tangled Tree, de David Quammen; livro-texto Biologia de Campbell, décima edição. Agradecemos Regina Lúcia Baldini, do Depto. de Bioquímica do IQ-USP, pela consultoria.
Os genes realmente egoístas: o que é o DNA lixo, a maior parte do nosso genoma Publicado primeiro em https://super.abril.com.br/feed








Nenhum comentário:
Postar um comentário